IGV バージョン 2.3 を立ち上げると、デフォルトでは Human hg18 のゲノムがデフォルトで表示される。その他のバージョンや生物種のゲノムを利用したい場合はサーバーからあるいはファイルからそのデータをインポートすることができる。
サーバーからリファレンスゲノムをインポートする
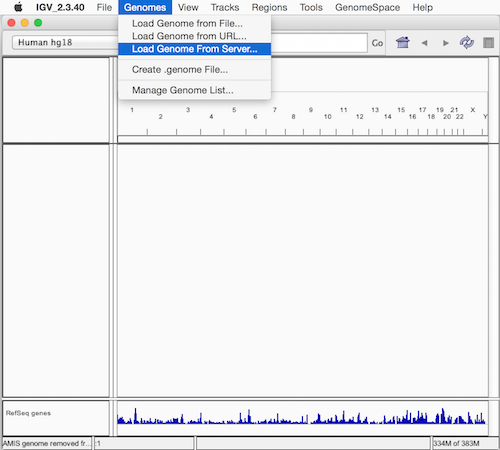
例えば、リファレンスゲノムのバージョンは生物種を変更したい場合は、igv のメニューバーから「Genomes」>「Load Genome From Server ...」を選択するとリファレンスゲノムの一覧が表示される。
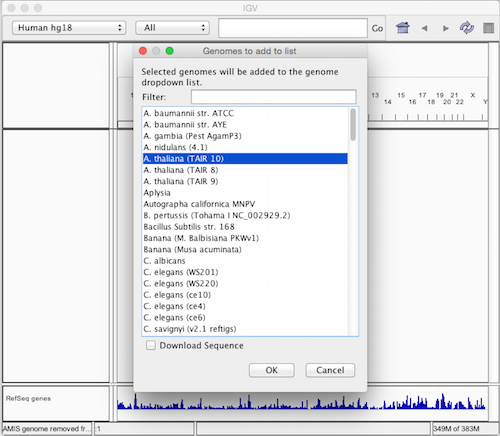
選択肢から利用したリファレンスゲノムを選択し、「OK」をクリックするとデータがダウンロードされる。
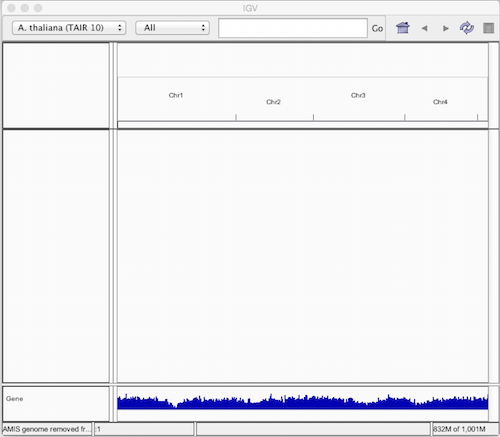
データのダウンロードが終了すると、IGV に選択したゲノムが表示される。この例では A.thaliana (TAIR 10) が表示される。
ファイルからリファレンスゲノムをインポートする
サーバーからインポートされるリファレンスゲノムは、ヒト、マウス、ラット、大腸菌などのように生物実験でよく使われている生物種に限られる。サーバーにないデータをインポートする場合は、予めリファレンスゲノムと GTF ファイルを Ensembl でダウンロードするか自分で準備する必要がある。
例えば、乳酸菌 12A のデータを以下のようにダウンロードできたとする。
- Lactobacillus_casei_12a.GCA_000309565.1.22.dna.toplevel.fa FTP
- Lactobacillus_casei_12a.GCA_000309565.2.24.gff3 FTP
IGV のメニューから「Genome > Create .genome file...」を選択する。
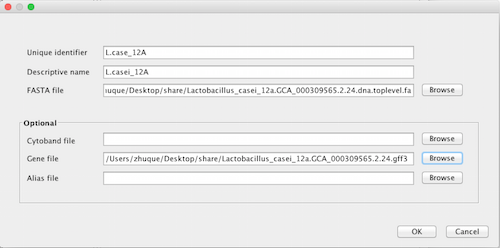
Unique identifier と Descriptive name にはゲノムの名前などを記入する。FASTA の欄にはダウンロードした toplevel の FASTA ファイルを指定する。Optional の Gene file にはダウンロードした GFF ファイルを指定する。
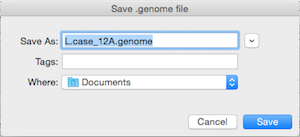
最後に .genome ファイルを保存する。
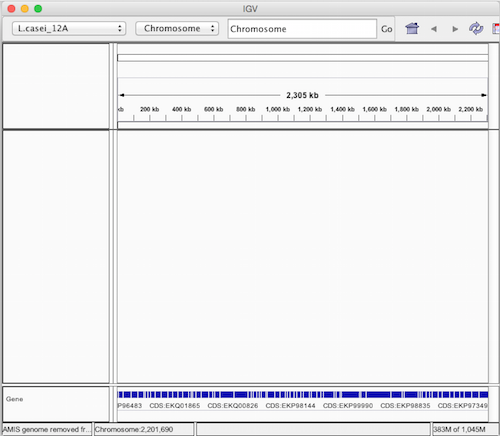
すると、作成したゲノムが読み込まれ、IGV にデータが表示される。表示範囲を拡大すると、GFF で定義された遺伝子名などが表示される。