シーケンサーから得られる FASTQ ファイルは、一般的に論文発表時あるいはその前に DDBJ SRA、NCBI SRA、EMBL-EBI ENA のいずれかのデータベースに登録される。論文中に記載される accession number を元にこれらのデータベースで調べれば、論文の解析に用いたデータを入手することができる。例えば、Blekhman らの論文では次のようにデータの登録番号を示してある。

データの登録番号は GSE で始まるものの他に、SRA や SRR で始まるものもある。いずれの場合に置いても、調べていくと SRR で始まる登録番号にたどり着く。例えば、上のように GSE17274 で記載されている場合、まず Google で検索するか、あるいは論文中に示された URL にアクセスする。検索結果からデータの公開ページにアクセスし、ページの下の方に SRA の登録番号(SRP001558)が見つかる(検索結果)。
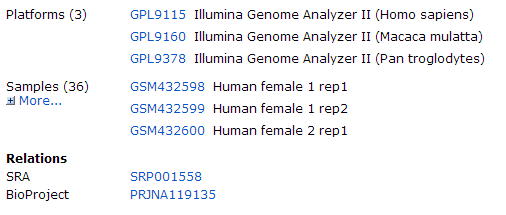
Relations には SRA の accession number が記述されている。SRP001558 のリンクをクリックすると、NCBI SRA のページに遷移される(NCBI SRA)。
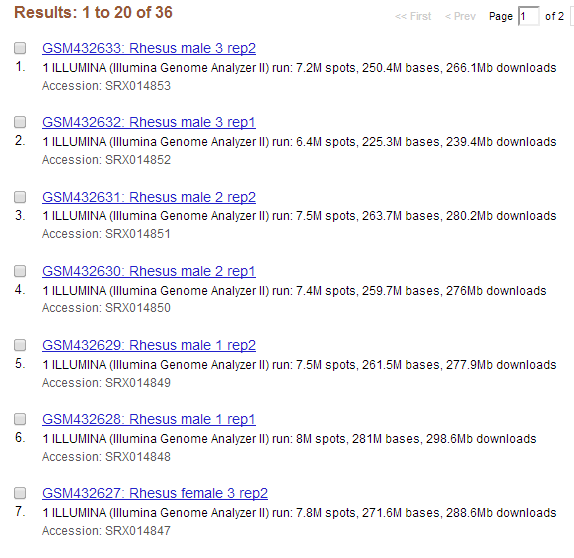
例えば「GSM432633: Rhesus male 3 rep2」リンクをクリックすると、以下のようなページが表示される(NCBI SRA)。
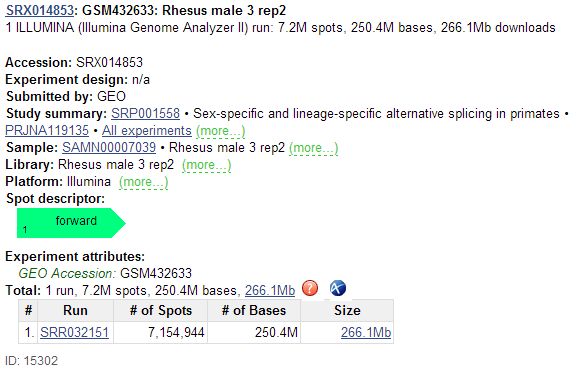
※ SRA の圧縮効率が上がったのか現在(2015年5月)では Size が 94Mb となっている。
ページの中 SRR の accession number がまとめられている表が見られる。例えば、SRR032151 の横の 266.1Mb のリンクをクリックするとダウンロードページに遷移される(ダウンロードページ)。ダウンロードページで SRR032151.sra をクリックすればダウンロードできるようになる。

ここでは例として、ヒトに関連するデータをすべてダウンロードする。ヒトに関連するデータの登録番号は以下のようになっている。
| GSM432598 Human female 1 rep1 | SRX014818 | SRR032116 | GSM432599 Human female 1 rep2 | SRX014819 | SRR032117 |
| GSM432600 Human female 2 rep1 | SRX014820 | SRR032118 | GSM432601 Human female 2 rep2 | SRX014821 | SRR032119 |
| GSM432602 Human female 3 rep1 | SRX014822 | SRR032120 | GSM432603 Human female 3 rep2 | SRX014823 | SRR032121 |
| GSM432604 Human male 1 rep1 | SRX014824 | SRR032122 | GSM432605 Human male 1 rep2 | SRX014825 | SRR032123 |
| GSM432606 Human male 2 rep1 | SRX014826 | SRR032124 | GSM432607 Human male 2 rep2 | SRX014827 | SRR032125 |
| GSM432608 Human male 3 rep1 | SRX014828 | SRR032126 | GSM432609 Human male 3 rep2 | SRX014829 | SRR032127 |
これらのデータはサイズが大きく、ファイル数も多いため、ここでは Linux のコマンド wget を利用してダウンロードする。
wget -c ftp://ftp-trace.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/SRR032/SRR032116/SRR032116.sra
wget -c ftp://ftp-trace.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/SRR032/SRR032117/SRR032117.sra
wget -c ftp://ftp-trace.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/SRR032/SRR032118/SRR032118.sra
wget -c ftp://ftp-trace.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/SRR032/SRR032119/SRR032119.sra
wget -c ftp://ftp-trace.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/SRR032/SRR032120/SRR032120.sra
wget -c ftp://ftp-trace.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/SRR032/SRR032121/SRR032121.sra
wget -c ftp://ftp-trace.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/SRR032/SRR032122/SRR032122.sra
wget -c ftp://ftp-trace.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/SRR032/SRR032123/SRR032123.sra
wget -c ftp://ftp-trace.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/SRR032/SRR032124/SRR032124.sra
wget -c ftp://ftp-trace.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/SRR032/SRR032125/SRR032125.sra
wget -c ftp://ftp-trace.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/SRR032/SRR032126/SRR032126.sra
wget -c ftp://ftp-trace.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/SRR032/SRR032127/SRR032127.sraこれらのデータは SRA と呼ばれるフォーマットで圧縮されているため、SRA toolkit 中の fastq-dump コマンドを利用して FASTQ を抽出する。
fastq-dump ./SRR032116.sra
fastq-dump ./SRR032117.sra
fastq-dump ./SRR032118.sra
fastq-dump ./SRR032119.sra
fastq-dump ./SRR032120.sra
fastq-dump ./SRR032121.sra
fastq-dump ./SRR032122.sra
fastq-dump ./SRR032123.sra
fastq-dump ./SRR032124.sra
fastq-dump ./SRR032125.sra
fastq-dump ./SRR032126.sra
fastq-dump ./SRR032127.sraスクリプトが正しく実行されると、ディレクトリには SRR032116.fastq ~ SRR032127.fastq ファイルが生成される。これが後の解析に利用する FASTQ ファイルである。