クラスタリングはサンプル同士の関係の調べるときによく利用される方法である。RNA-Seq 解析では、一般に複数のサンプルに対してシークエンシングが行われる。ストレス処理群と対照群の比較実験であれば、処理群 3 サンプル、対照群 3 サンプルからなる実験が多い。また、組織別で処理群と対照群の比較実験であれば、サンプル数は組織の数だけ増える。さらに、時系列実験であれば、数十サンプルから数百サンプルに及ぶ場合もある。実際の発現量解析を行う前に、これらのサンプルの特徴を把握していくことが重要である。各サンプルの特徴を把握して初めて、実験結果と計画がどれぐらいかけ離れているのかを確認することができるようになり、計画を修正したり、あるいは新しい発見を計画に加えたりすることができるようになる。
サンプル間の特徴を調べたり、サンプル同士の類似度を調べるために階層的クラスタリング、k-means、主成分分析(PCA) がよく使われている。そのほかに、多次元尺度構成法(MDS)や非負行列分解などの方法も挙げられるが、ほとんど使われていない。
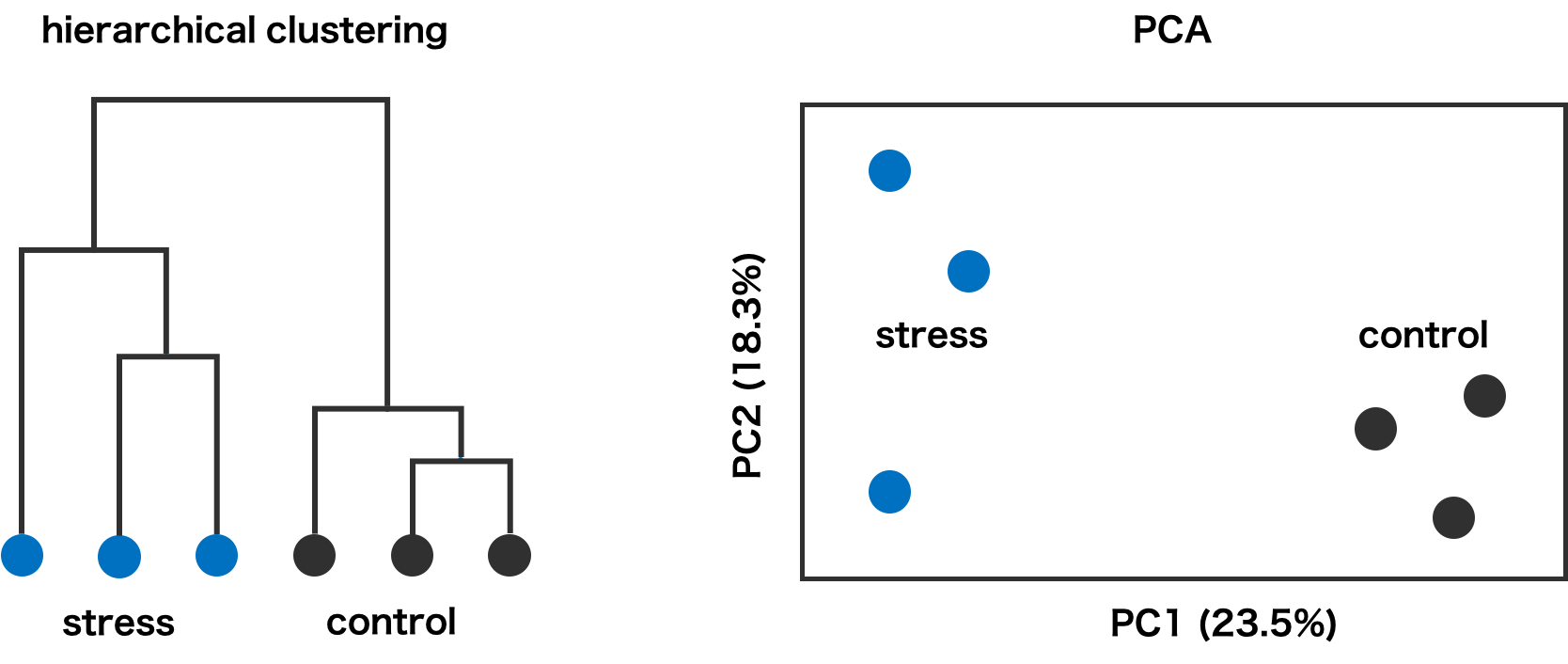
解析を行う前にクラスタリングを行うことで、大まかな結果を見積もったり、間違っていることを早く発見できたりする。例えば、ストレス処理群と対照群の比較実験であれば、クラスタリングで処理群のサンプルと対照群のサンプルが互いに異なるクラスターに属していることを確認する必要がある。クラスタリング結果で両者のクラスター間の距離が小さければ、処理群と対照群の間に大きな差異がないと考えられる。このとき、検出できる発現変動遺伝子の数も少ないと思われる。また、処理群のクラスターの中に対照群のサンプルが 1 つだけ入り混じっているようであれば、サンプルの取り違えなどの可能性が高くなる。特にサンプル数の多い、組織別の実験や時系列実験の場合は、実験過程のヒューマンエラーも多くなりがちで、必ずクラスタリング結果を確認すべきである。
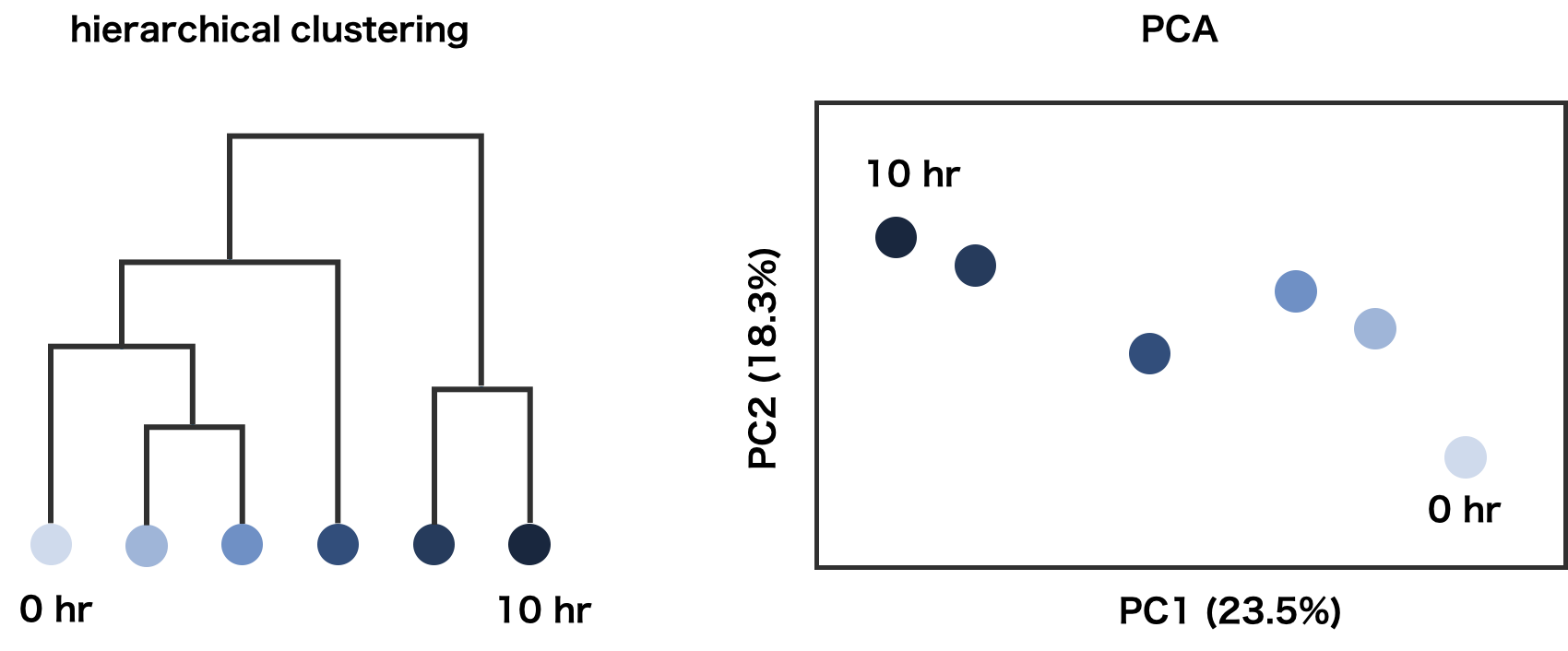
時系列データの場合、時系列的な変化が見られる場合が多い。このとき、階層的クラスタリングや PCA の結果からは、時間経過を表す軌跡のような傾向が見えることが多い。