PDB には様々な便利なウェブインターフェイスが用意されている(PDB RESTful Web Service interface)。これらのインターフェイスを利用することで、Python からクエリーを作成して PDB に対してホモロジー検索を行うことができる。手順としては、(1) XML フォーマットのクエリーを作成する、(2) urllib モジュールを利用して、クエリーを送信し、結果を取得するの 2 ステップからなる。
クエリーの作成
簡単なクエリーのサンプルは、PDB RESTful Web Service interface のページで確認できる。例えば、Accession ID が P50225 であるタンパク質を検索したい場合は次のようにする。
<orgPdbQuery>
<queryType>org.pdb.query.simple.UpAccessionIdQuery</queryType>
<accessionIdList>P50225</accessionIdList>
</orgPdbQuery>また、複雑なクエリーを作成する場合は、まず、PDB の advanced search ページで一度検索する。すると、検索結果のページでは Query details のリンクが表示されるので、このリンク先でクエリー文を確認できる。
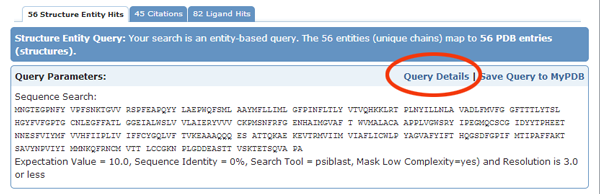
クエリーの送信と結果の取得
クエリーを作成したら、次に PDB に送信し検索を行う。このとき、Python の urllib モジュールを利用する(Python 2.x の場合は urllib2 を利用する)。クエリーは http://www.rcsb.org/pdb/rest/search に送信する必要がある。
次は、Accession ID が P50225 とアノテーションされているタンパク質を検索する例である。
import urllib
import urllib.request
url = 'http://www.rcsb.org/pdb/rest/search'
query = '''
<orgPdbQuery>
<queryType>org.pdb.query.simple.UpAccessionIdQuery</queryType>
<accessionIdList>P50225</accessionIdList>
</orgPdbQuery>
'''
req = urllib.request.Request(url, data = query.encode('ascii'))
fet = urllib.request.urlopen(req)
fet.read().decode('utf-8')
## '1LS6\n1Z28\n2D06\n3QVU\n3QVV\n3U3J\n3U3K\n3U3M\n3U3O\n3U3R\n4GRA\n'BLAST 検索
BLAST 検索や複数条件による検索も行える。クエリーの作成が難しいため、まず一度 advanced search で検索して、雛形となるクエリーを取得する。次に Python でこのクエリーを編集するなりして、自分のほしいクエリーを作成する。
次の Python スクリプトは、seq 配列をクエリーとして、PDB に対して PSI-BLAST 検索を行い、検索結果から 3 Å 以下の解像度の X 線結晶解析によって構造決定されたタンパク質で、かつ、e-value が 1.0 よりも小さいタンパク質を結果として出力している。
import urllib
import urllib.request
pdb_req_url = 'http://www.rcsb.org/pdb/rest/search'
seq = """
MNGTEGPNFYVPFSNKTGVVRSPFEAPQYYLAEPWQFSMLAAYMFLLIMLGFPINFLTLYVTVQHKKLRTPLNYILLNLA
VADLFMVFGGFTTTLYTSLHGYFVFGPTGCNLEGFFATLGGEIALWSLVVLAIERYVVVCKPMSNFRFGENHAIMGVAFT
WVMALACAAPPLVGWSRYIPEGMQCSCGIDYYTPHEETNNESFVIYMFVVHFIIPLIVIFFCYGQLVFTVKEAAAQQQES
ATTQKAEKEVTRMVIIMVIAFLICWLPYAGVAFYIFTHQGSDFGPIFMTIPAFFAKTSAVYNPVIYIMMNKQFRNCMVTT
LCCGKNPLGDDEASTTVSKTETSQVAPA
"""
params = {
'q_seq' : seq,
'q_tool' : 'psiblast',
'q_e_cutoff' : 1.0,
'q_identity_cutoff' : 30,
'q_max_resolution' : 3.0
}
xml_query = """
<orgPdbCompositeQuery version="1.0">
<queryRefinement>
<queryRefinementLevel>0</queryRefinementLevel>
<orgPdbQuery>
<version>head</version>
<queryType>org.pdb.query.simple.SequenceQuery</queryType>
<sequence>%(q_seq)s</sequence>
<searchTool>%(q_tool)s</searchTool>
<eValueCutoff>%(q_e_cutoff)s</eValueCutoff>
<sequenceIdentityCutoff>%(q_identity_cutoff)s</sequenceIdentityCutoff>
</orgPdbQuery>
</queryRefinement>
<queryRefinement>
<queryRefinementLevel>1</queryRefinementLevel>
<conjunctionType>and</conjunctionType>
<orgPdbQuery>
<version>head</version>
<queryType>org.pdb.query.simple.ResolutionQuery</queryType>
<refine.ls_d_res_high.comparator>between</refine.ls_d_res_high.comparator>
<refine.ls_d_res_high.max>%(q_max_resolution)s</refine.ls_d_res_high.max>
</orgPdbQuery>
</queryRefinement>
</orgPdbCompositeQuery>
""" % params
req = urllib.request.Request(pdb_req_url, data = xml_query.encode('ascii'))
fet = urllib.request.urlopen(req)
pdb_list = fet.read().decode('utf-8').split("\n")
fet.close()
pdb_list.pop()
print(pdb_list)
## ['1F88:1', '1GZM:1', '1HZX:1', '1L9H:1', '1U19:1', '2G87:1', '2HPY:1', '2PED:1', '2X72:1', '3C9L:1', '3CAP:1', '3OAX:1', '3PQR:1', '3PXO:1', '4BEY:1', '4J4Q:1', '4PXF:1', '4X1H:1', '5DYS:1', '5EN0:1', '5TE3:1']