予測したい配列を最初に二次構造予測を行い、その二次構造予測の結果を利用して三次構造を予測することができる。ここでは、3PQR の構造を例として取り上げて、構造を予測する。3PQR の A 鎖のアミノ酸配列は以下のようになっている。
>3PQR:A|PDBID|CHAIN|SEQUENCE MNGTEGPNFYVPFSNKTGVVRSPFEAPQYYLAEPWQFSMLAAYMFLLIMLGFPINFLTLYVTVQHKKLRTPLNYILLNLA VADLFMVFGGFTTTLYTSLHGYFVFGPTGCNLEGFFATLGGEIALWSLVVLAIERYVVVCKPMSNFRFGENHAIMGVAFT WVMALACAAPPLVGWSRYIPEGMQCSCGIDYYTPHEETNNESFVIYMFVVHFIIPLIVIFFCYGQLVFTVKEAAAQQQES ATTQKAEKEVTRMVIIMVIAFLICWLPYAGVAFYIFTHQGSDFGPIFMTIPAFFAKTSAVYNPVIYIMMNKQFRNCMVTT LCCGKNPLGDDEASTTVSKTETSQVAPA
次に、PSI-BLAST をかけてテンプレートを探す。この配列と同じ構造が複数登録されているため、検索結果に 100% 一致するような配列が含まれる。それらの配列を避けて使わないことにする。ここでは、2ZIY を利用する。2ZIY と 3PQR(TARGET)のアラインメントを以下のように整理し、これを alignments.ali の名前で保存する。
>P1;TARGET sequence:TARGET:::::::: MNGTEGPNFYVPFSNKTGVVRSPFEAPQYYLAEPWQFSMLAAYMFLLIMLGFPINFLTLY VTVQHKKLRTPLNYILLNLAVADL-FMVFGGFTTTLYTSLHGYFVFGPTGCNLEGFFATL GGEIALWSLVVLAIERYVVVCKPMS-NFRFGENHAIMGVAFTWVMALACAAPPLVGWSRY IPEGMQCSCGIDYYTPHEETNNESFVIYMFVVHFIIPLIVIFFCYGQLVFTV----KEAA A--------QQQESATTQKAEKEVTRMVIIMVIAFLICWLPYAGVAFYIFTHQGSDFGPI ------FMTIPAFFAKTSAVYNPVIYIMMNKQFRNCMVTTL-----CC* >P1;2ZIY structureX:2ZIY:57:A:337:A:::: -----------------------------------------------------------Y LFTKTKSLQTPANMFIINLAFSDFTFSLVNGFPLMTISCFLKKWIFGFAACKVYGFIGGI FGFMSIMTMAMISIDRYNVIGRPMAASKKMSHRRAFIMIIFVWLWSVLWAIGPIFGWGAY TLEGVLCNCSFDYIS--RDSTTRSNILCMFILGFFGPILIIFFCYFNIVMSVSNHEKEMA AMAKRLNAKELRKAQAGANAEMRLAKISIVIVSQFLLSWSPYAVVALL------AQFGPL EWVTPYAAQLPVMFAKASAIHNPMIYSVSHPKFREAISQTFPWVLTCC*
次に、Jpred を利用して、3PQR(TARGET)の二次構造を予測する。予測結果から、以下に示す領域に α-ヘリックスが存在することがわかったとする。
MNGTEGPNFYVPFSNKTGVVRSPFEAPQYYLAEPWQFSMLAAYMFLLIMLGFPINFLTLY -----------------------------------HHHHHHHHHHHHHHHHHHHHHHHHE VTVQHKKLRTPLNYILLNLAVADLFMVFGGFTTTLYTSLHGYFVFGPTGCNLEGFFATLG EEEE-------HHHHHHHHHHHHHHHHHHHHHHHHHHHH--------HHHHHHHHHHHHH GEIALWSLVVLAIERYVVVCKPMSNFRFGENHAIMGVAFTWVMALACAAPPLVGWSRYIP HHHHHHHHHHHHHHHHHHE-----------HHHHHHHHHHHHHHHHHHHHHHHHHHHH-- EGMQCSCGIDYYTPHEETNNESFVIYMFVVHFIIPLIVIFFCYGQLVFTVKEAAAQQQES ---------------HHHHHHHHHHHHHHHHHHHHHHHHHHHHHHHHHHHHHHHHHH--- ATTQKAEKEVTRMVIIMVIAFLICWLPYAGVAFYIFTHQGSDFGPIFMTIPAFFAKTSAV --HHHHHHHHHHHHHHHHHHHHHHHHHHHHHHHHH---------HHHHHHHHHHHHHHH- YNPVIYIMMNKQFRNCMVTTLCCGKNPLGDDEASTTVSKTETSQVAPA --HHHHEE--HHHHHHHHHHHH-----------------EEEE-----
モデリング用のスクリプトを作成する。Jpred の予測結果を参考にして、α ヘリックスと予測された領域を secondary_structure.alpha で α へリックにモデリングさせる。
from modeller import *
from modeller.automodel import * # Load the automodel class
log.verbose()
env = environ()
env.io.atom_files_directory = ['.']
class MyModel(automodel):
def special_restraints(self, aln):
rsr = self.restraints
at = self.atoms
# alpha helix
rsr.add(secondary_structure.alpha(self.residue_range('36:', '59:')))
rsr.add(secondary_structure.alpha(self.residue_range('72:', '99:')))
rsr.add(secondary_structure.alpha(self.residue_range('108:', '138:')))
rsr.add(secondary_structure.alpha(self.residue_range('151:', '178:')))
rsr.add(secondary_structure.alpha(self.residue_range('196:', '237:')))
rsr.add(secondary_structure.alpha(self.residue_range('243:', '275:')))
rsr.add(secondary_structure.alpha(self.residue_range('285:', '299:')))
rsr.add(secondary_structure.alpha(self.residue_range('303:', '306:')))
rsr.add(secondary_structure.alpha(self.residue_range('311:', '322:')))
a = MyModel(env,
alnfile = 'alignments.ali',
knowns = '2ZIY',
sequence = 'TARGET')
a.starting_model= 1
a.ending_model = 1
a.make()スクリプトを predict_target.py として保存して、MODELLER で実行すると、予測結果として TARGET.B99990001.pdb ファイルが生成される。(テンプレートの 2ZIY の PDB ファイルを予め、ダウンロードしておく必要がある。)
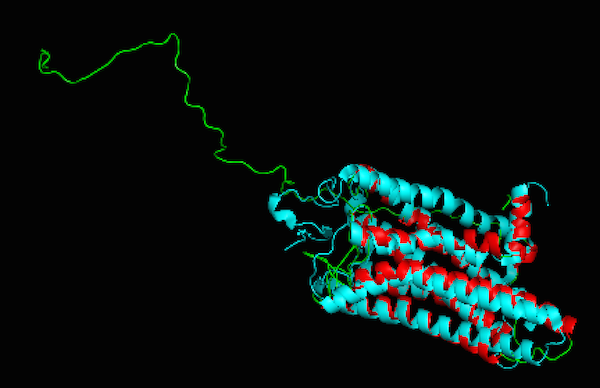
mod9.13 predict_target.py予測した構造(ヘリックスが赤)と実際の構造(ヘリックスが水色)を重ね合わせてみると、以下のようになる。N 末端側の disorder 領域と、N 末端最初のヘリックスは予測構造と実際構造は、テンプレートがないためだったか、大きくずれている。それ以外の部分はおおむねよく予測されている。
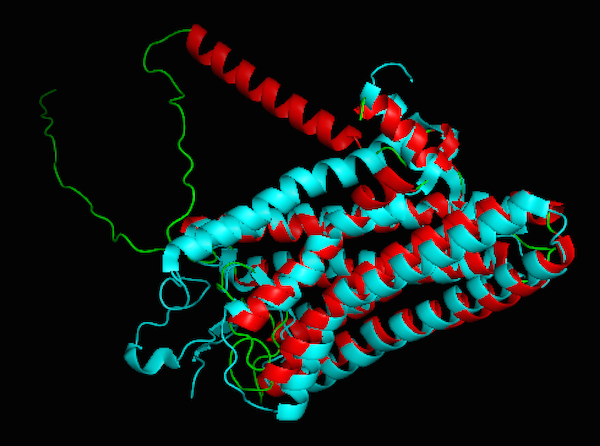
secondary_structure 関数を利用しないで、予測すると、以下のような結果となる。N 末端の最初の α-ヘリックスがうまく予測されていない。