MODELLER の secondary_structure.alpha と secondary_structure.strand メソッドを組み合わせることで、α-ヘリックスと β-シートが組合わさった二次構造を作成することができる。
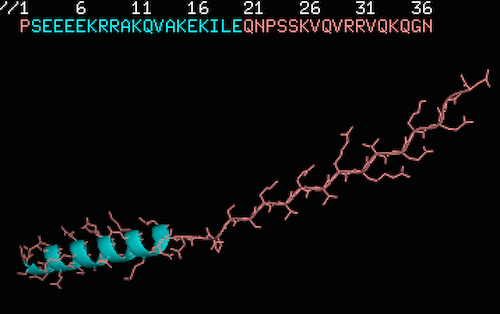
作成手順としては、以下のようにステップ分けできる。例に用いる一次配列を、4R80 の一部「PSEEEEKRRAKQVAKEKILEQNPSSKVQVRRVQKQGN」とする。実際のタンパク質の中では、2-20 番目のアミノ酸は α-ヘリックス、23-35 番目のアミノ酸は β-ストランドとなっている。
- 一次配列のモデルを作成する(build_sequence)
- タンパク質が取りうる構造を計算する(restraints.make)
- 二次構造を作成する
(secondary_structure.alpha と secondary_structure.strand) - 構造を最適化する(optimize)
Python スクリプトは以下のようになる。
from modeller import *
from modeller.optimizers import conjugate_gradients
env = environ()
env.libs.topology.read('${LIB}/top_heav.lib')
env.libs.parameters.read('${LIB}/par.lib')
# 1.
m = model(env)
# 1234567890123456789012345678901234567
# HHHHHHHHHHHHHHHHHHH EEEEEEEEEEEEE
m.build_sequence('PSEEEEKRRAKQVAKEKILEQNPSSKVQVRRVQKQGN')
# 2.
allatoms = selection(m)
m.restraints.make(allatoms, restraint_type='STEREO', spline_on_site=False)
# 3.
m.restraints.add(secondary_structure.alpha(m.residue_range('2:', '20:')))
m.restraints.add(secondary_structure.strand(m.residue_range('23:', '35:')))
# 4.
cg = conjugate_gradients()
cg.optimize(allatoms, max_iterations = 10000)
# save to PDB format
m.write(file='alphabetastrand.pdb')Python スクリプトを make_alphabetastrand.py として保存し、MODELLER で実行すれば、結果として alphabetastrand.pdb ファイルが生成される。
mod9.13 make_alphabetastrand.py作成されたモデルを PyMOL で見ると、以下のようになる。