LOMETS (Wu et al., 2007) は内部的に MUSTER などのプログラムを利用して、最適なテンプレートを探し、テンプレートに基づいてタンパク質の立体構造をモデリングするプログラムである。このプログラムは、Zhang Lab のウェブサイトで直接利用できるほか、I-TASSER Suit をダウンロードしてローカルでも利用できる。
ローカルで利用する場合
LOMETS は I-TASSER Suit にパッケージングされている。I-TASSER Suit をインストールすれば、runLOMETS.pl スクリプトを実行すればよい。
例として、以下の配列の立体構造をモデリングしてみる。
>T0800 MNIKKLFSIFTLVILVFATACTNKSEKATVSDTNEKPKEEIKIVEPNGAEKTKLNLNFGVGKLNISGN EEKLMKGKFIYSENEWKPEIKYEVKDKDGELEISQPGLKSGNVSLNNKRNEWNINLNEKIPTEIKLSL GTGEFKADLSKINLKELNVGMGVGKVDLDISGNYKNNVKVNIEGGVGEATVYLPKSIGVKIKAEKGVG AVNANGFIVEGENIYKNSQYGKSKNSIEVNIEAGVGAINIKQK
この配列を seq.fasta の名前で保存する。次に、この seq.fasta ファイルを targets ディレクトリに保存して準備完了。最後に I-TASSER Suit 中の runLOMETS.pl を実行すればよい。モデリングに要する時間は非常に長いため、ここでは nohup を用いて実行例を示した。
nohup runLOMETS.pl -pkgdir /tools/I-TASSER4.1 \
-libdir /tools/I-TASSER4.1/lib \
-seqname T0800 \
-datadir ./lomets \
-java_home /usr &オプションとして、-pkgdir は I-TASSER Suit のインストールディレクトリ、-libdir は I-TASSER Suit 用のライブラリーを指定する。-seqname には予測する配列の名前、-datadir には seq.fasta が保存されているディレクトリを指定する。最後に -java_home には JAVA_HOME のパスを指定する。Linux であればほとんどの場合 /usr となる。
ウェブサイトで利用する場合
Zhang lab のウェブページにいけば、ウェブベースの LOMETS を利用することができる。ページ中のフォームに予測したい配列を入力し、E-Mail アドレスおよびタスク ID を入力し、「Run LOMETS」ボタンをクリックすればよい。実行結果はメールで知らせてくれる。
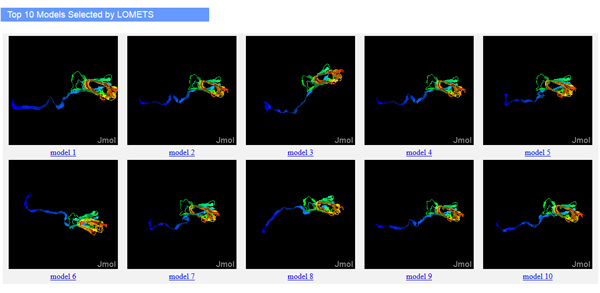
メールで送られるリンクをクリックすれば以下の様なページが表示される。それぞれのモデルをクリックすれば、PDB ファイルをダウンロードできる。
References
- LOMETS: a local meta-threading-server for protein structure prediction. Nucleic Acids Res. 2007, 35(10)3375-82. DOI: 10.1093/nar/gkm251 PMID: 17478507