ある遺伝子について考えたとき、その遺伝子の持つ機能や細胞中における局在場所などにおいて、様々な視点aから見ることができる。着目点が異なれば、その捉え方も異なってくる。つまり、場合によっては同じ遺伝子を取り扱っていながら、違う機能あるいは違う名前で呼ばれたりして、あたかも違う遺伝子であるかのように取り扱われる場合が多い。そこで、このようなことが起こらないように遺伝子オントロジーが考えられ、GO が定義されている。GO は常に最新の生物学的な知識を反映させるために、毎日更新を行っている。また、最新の知見で、先行研究が否定されたような場合は GO の変更や削除なども行われている。
現在までにおよそ 4 万個の GO が定義されている。例えば、新しい遺伝子について研究し、ある程度まとまれば、その遺伝子を記述するためには、この 4 万個の GO の中から最適なものを探し、その遺伝子を記述する。すべての GO は、次に挙げた 3 つのカテゴリーのうちどれか 1 つだけに分類される。
- biological process(生物学的プロセス)
- cellular component(細胞の構成要素)
- molecular function(分子機能)
1 つの遺伝子に複数の GO が付けられたり、1 個の GO が複数の遺伝子を記述できたりする。このように 1 つの GO と 1 つの遺伝子の間に関係を結ぶことをアノテーションという。現在までには 4 万個の GO に対して、アノテーション数は 2 億に上る。この内、1% が manual annotations であり、それ以外は automatic annotations である。manual annotations は主にスタッフが論文を読んで、論文を裏付け情報として GO と遺伝子間のアノテーションを行っている。およそ 25 万個のタンパク質が manual annotations を持つ。一方、automatic annotations は主に、オーソログ、タンパク質ドメイン、配列類似度などを元に機械的にアノテーションを行っている。
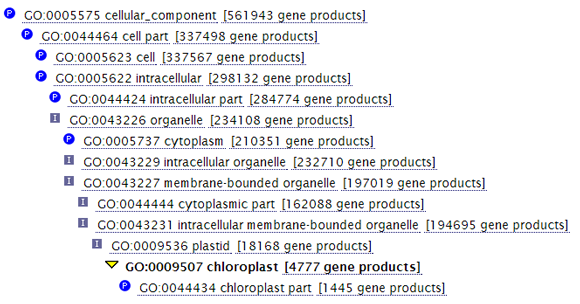
GO は階層構造を持つために、その遺伝子に定義された GO に親階層が存在すると、その親階層もその遺伝子の GO と定義される。例えば、次の図で確認できるように、ある遺伝子に関して「chloroplast」が定義されていると、その親・祖先にあたる「cytoplasm」、「intracellular part」なども自動的に定義される。
GO は様々な方法で付けられる。例えば、in vitro 実験結果に基づいてつけたり、塩基配列相同性に基づいてつけたり、系統解析の結果を考慮して付けたりしている。GO がどのように付けられたのかを示すために、evidence code が用意されている。
以下に示す evidence code は experimental evidence code であり、主に実験結果を参考にして GO が付けられたことを意味する。Gene Ontology のサイトで実例とともに詳しい解説がなされている。
| code | 定義と例 |
| EXP | Inferred from Experiment アノテーションが実験結果をもとに付けられていることを意味する。EXP は IDA、IPI、IMP、IGI および IEP の親コードにあたる。 |
| IDA | Inferred from Direct Assay 酵素アッセイ、in vitro、免疫蛍光抗体法、細胞破砕法など。 |
| IPI | Inferred from Pyhsical Interaction ツーハイブリッド法、共免疫沈降法など。 |
| IMP | Inferred from Mutant Phenotype ノックアウト、遺伝子破壊、遺伝子導入、RNAi、anti-sense RNA など。野生型と比較して遺伝子の機能や局在などを決定した場合に付けられる。 |
| IGI | Inferred from Genetic Interaction 遺伝子相互作用、ノックアウト、遺伝子破壊、遺伝子導入法など。複数のノックアウト型を比較したりして遺伝子機能を決定した場合は(IMP ではなく) IGI を利用する。 |
| IEP | Infrred From Expression Pattern マイクロアレイによるトランスクリプトーム解析、ウェスタンブロッティングなど。 |
次の evidence code は computational analysis evidence code であり、バイオインフォマティクス知識によって付けられたことを意味する。
| code | 定義と例 |
| ISS | Inferred from Sequence or Structural Similarity 塩基(あるいはアミノ酸)配列の特徴に基いて決定した GO に付けられる。ISA、ISO および ISM の親コードにあたる。 |
| ISO | Inferred from Sequence Orthology 配列アライメント。 |
| ISA | Inferred from Sequence Alignment 他の生物種の遺伝子のオントロジーを参照して決定した場合に付けられる。 |
| ISM | Inferred from Sequence Model HMM なども配列モデリングツールによって決定した場合に付けられる。 |
| IGC | Inferred from Genomic Context パスウェイ解析、遺伝子網羅的解析など。 |
| IBA | Inferred from Biological aspect of Ancestor 系統学的に祖先の機能を参照して決定された場合に付けられる。 |
| IBD | Inferred from Biological aspect of Descendant 系統学的に子孫の機能を参照して決定された場合に付けられる。 |
| IKR | Inferred from Key Residues |
| IRD | Inferred from Rapid Divergence |
| RCA | inferred from Reviewed Computational Analysis |
次のいくつかは author statement evidence code であり、論文著者らの提唱した内容に基いて付けられていることを意味する。
| code | 定義と例 |
| TAS | Traceable Author Statement 実験的に示されていないが、論文の中で取り上げられた。 |
| NAS | Non-traceable Author Statement 論文中に取り上げられているが、その論文以外では参照・引用がなされていない。 |
その他の evidence code。
| code | 定義と例 |
| IC | Inferred by Curator |
| ND | No biological Data available |
| IEA | Inferred from Electronic Annotation |
| NR | Not Recorded |
References
- Gene ontology: tool for the unification of biology. The Gene Ontology Consortium. Nat Genet. 2000, 25(1):25-9. PubMed Abstract
- Understanding how and why the Gene Ontology and its annotations evolve: the GO within UniProt. Gigascience. 2014, 3(1):4. PubMed Abstract