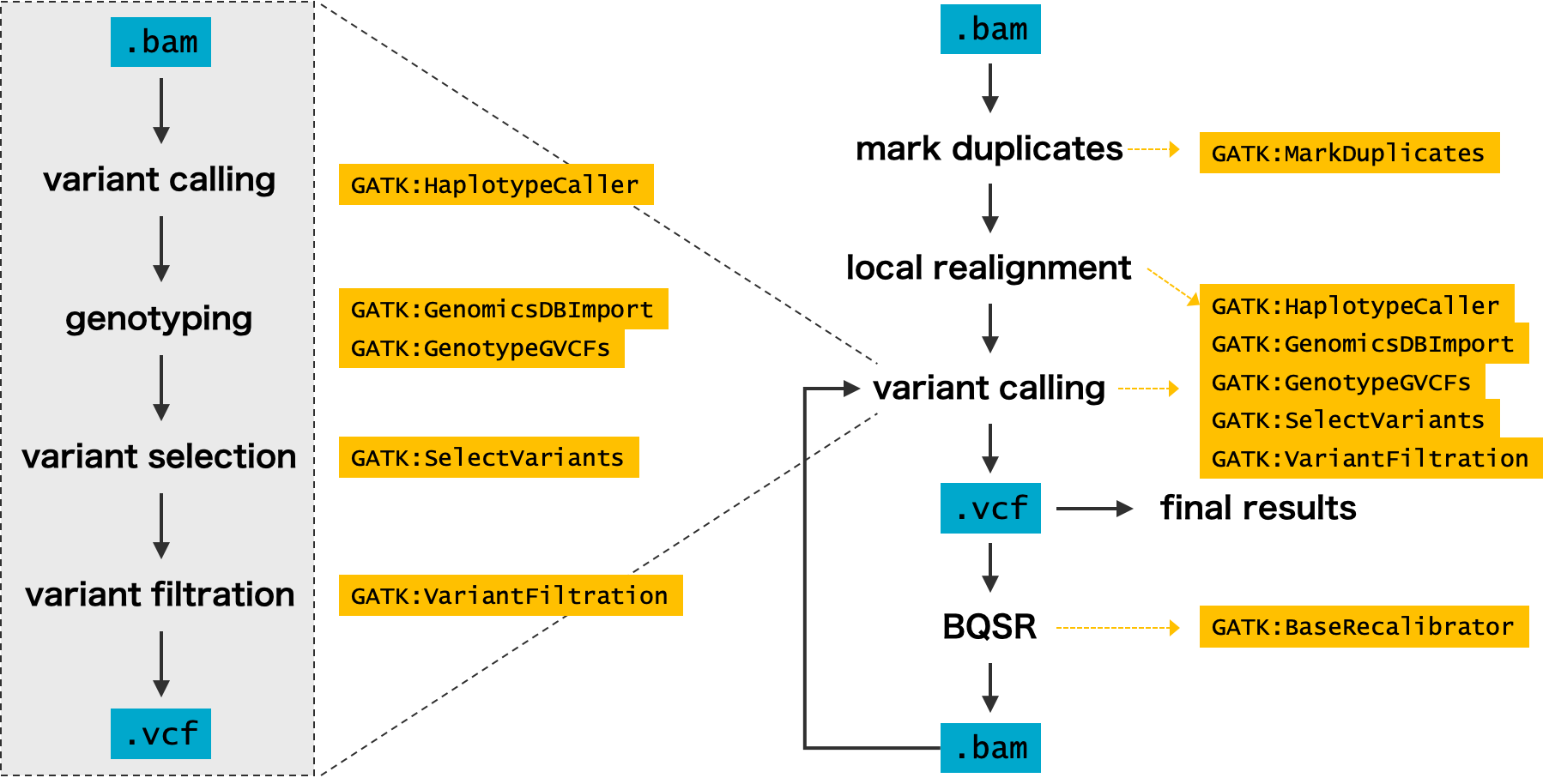
GATK は、シーケンサーデータからバリアントを検出するためのプログラム群である。Java で実装されている。主に使うコマンドを以下に示した。
| CreateSequenceDictionary | リファレンス配列の GATK 用のインデックスを作成するためのコマンド。 |
| MarkDuplicates | BAM ファイル中の重複リードに識別情報を追加するためのコマンド。 |
| CollectAlignmentSummaryMetrics | BAM ファイルに記録されたマッピング結果の統計量を計算するためのコマンドである。 |
| CollectInsertSizeMetrics | BAM ファイルに記録された paired-end リードの insertion size の分布を調べるためのコマンドである。 |
| BaseRecalibrator | BAM ファイル中にあるリードのクオリティスコアを補正するためのデータを計算するコマンドである。 |
| ApplyBQSR | BAM ファイル中のリードのクオリティスコアを補正するためのコマンドである。 |
| HaplotypeCaller | ハプロタイプを推定するためのコマンドである。 |
| CombineGVCFs | 複数の gVCF ファイルを一つの gVCF ファイルにマージするためのコマンドである。 |
| GenomicsDBImport | 複数の gVCF ファイルからジェノタイピング用のローカルデータベースを構築するためのコマンドである。 |
| GenotypeGVCFs | gVCF ファイルを利用してジェノタイピングを行うためのコマンドである。 |
| SelectVariants | VCF ファイルから特定のバリアントを抽出するためのコマンドである。 |
| VariantFiltration | 閾値を設けてバリアントをフィルタリングするためのコマンドである。 |