ATTED-II は植物の遺伝子共発現データベースである(Aoki et al., 2016)。ATTED-II version 11.0 では、実験でもっとも使われているシロイヌナズナをはじめとして、重要作物である菜の花、大豆、トマト、稲やトウモロコシなども含めて、計 9 植物種の共発現データベースが公開されている (Obayashi et al., 2022)。共発現データベースを活用することで、機能が似た遺伝子を同定できたり、ある遺伝子の上流あるいは下流にある遺伝子を同定できたり、さらには種間における遺伝子発現パターンの相違を調べたりすることができるようになる。
共発現データ
ATTED-II で公開されている共発現データは、NCBI GEO や EBI ArrayExpress などのデータベースで公開されているマイクロアレイおよび RNA-Seq のデータを解析して得られているものである。NCBI GEO や EBI ArrayExpress などで公開されているデータは、それぞれの研究目的のために取られたものが多い。例えば、植物の特定の組織の遺伝子発現量データだったり、植物に何らかのストレスを与える前と後の遺伝子発現量データだったり、あるいはストレスを与えた後の時系列発現量データだったりすることが多い。そのため、これらのデータをそのまま利用して共発現データを計算すると、共発現データが植物の平均的な状態を反映できない可能性が生じる。そこで、ATTED-II では、主成分分析を活用して、各実験に特異的なノイズを取り除き、condition-independent な遺伝子共発現データを計算している。
共発現データを計算方法は、ATTED-II データベースがアップデートするごとに改良が続けられている。version 11.0 では、遺伝子発現量データ(行列)に対して主成分分析を行い、最初の 1000 個の主成分に対してアンサンブル学習の一つである subagging で特徴抽出を行い(重要な主成分を抽出し)、その重要な主成分に対してピアソン相関係数を計算し標準化を行い z 値を出している。この z 値は標準化されているので、同データベースにある他の遺伝子や多種の植物との比較も可能である。
行列形式の共発現データは ATTED-II ダウンロードページの Coexpressed gene table リンクからダウンロードできる。また、行列データをダウンロードしなくても、以下のように、遺伝子名を入力して、共発現する遺伝子を手軽に検索できる。
共発現遺伝子の検索
共発現遺伝子を検索は ATTED-II CoExSearch で行う。例えば、エチレン受容体遺伝子 ERS1 (AT2G40940) の共発現遺伝子を調べるには、検索欄に遺伝子の TAIR ID を入力する。遺伝子名による検索は、現段階では未対応のようである。
検索結果は、次のように表形式で出力される。検索結果をみると、ERS1 と共発現する遺伝子には、EBF2、ERS2、ETR2、CTR1、RAP2.2、EIL1 などがある。ERS1 と共発現する遺伝子はエチレン応答経路上の遺伝子が多いことがわかる。なお、ATTED-II のバージョンによって出力が異なる場合がある。

共発現ネットワークの作成
ATTED-II の NetwordDrawer で共発現ネットワークを作成することができる。例えば、エチレン応答経路上のいくつかの遺伝子を利用して、共発現ネットワークを作成する場合は、以下のようにする。
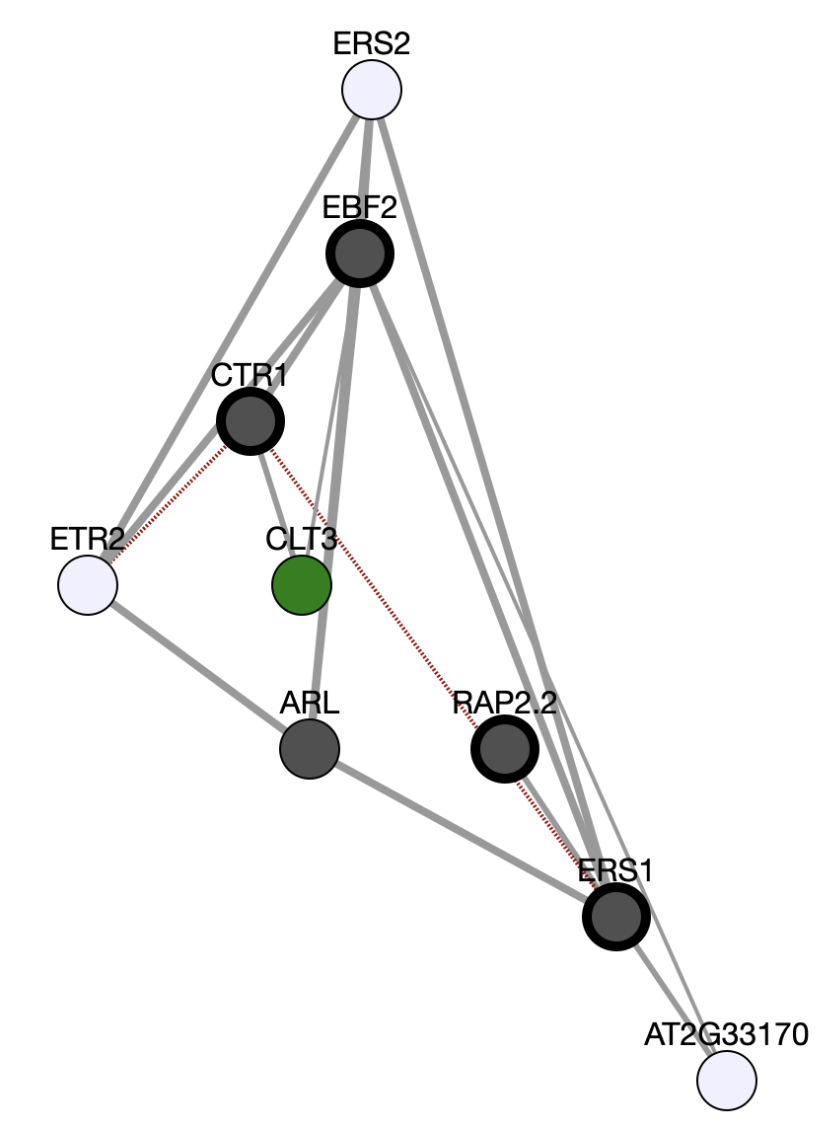
作成された共発現ネットワークは次のようになる。共発現ネットワーク上に、入力した遺伝子の他にもいくつかの遺伝子が追加されている。グラフの出力形式はいくつかの種類があり、出力画面で選ぶことができる。
References
- ATTED-II in 2016: A Plant Coexpression Database Towards Lineage-Specific Coexpression. Plant Cell Physiol. 2015, 57(1):e5. DOI: 10.1093/pcp/pcv165
- ATTED-II v11: A Plant Gene Coexpression Database Using a Sample Balancing Technique by Subagging of Principal Components. Plant Cell Physiol. 2022. 10.1093/pcp/pcac041